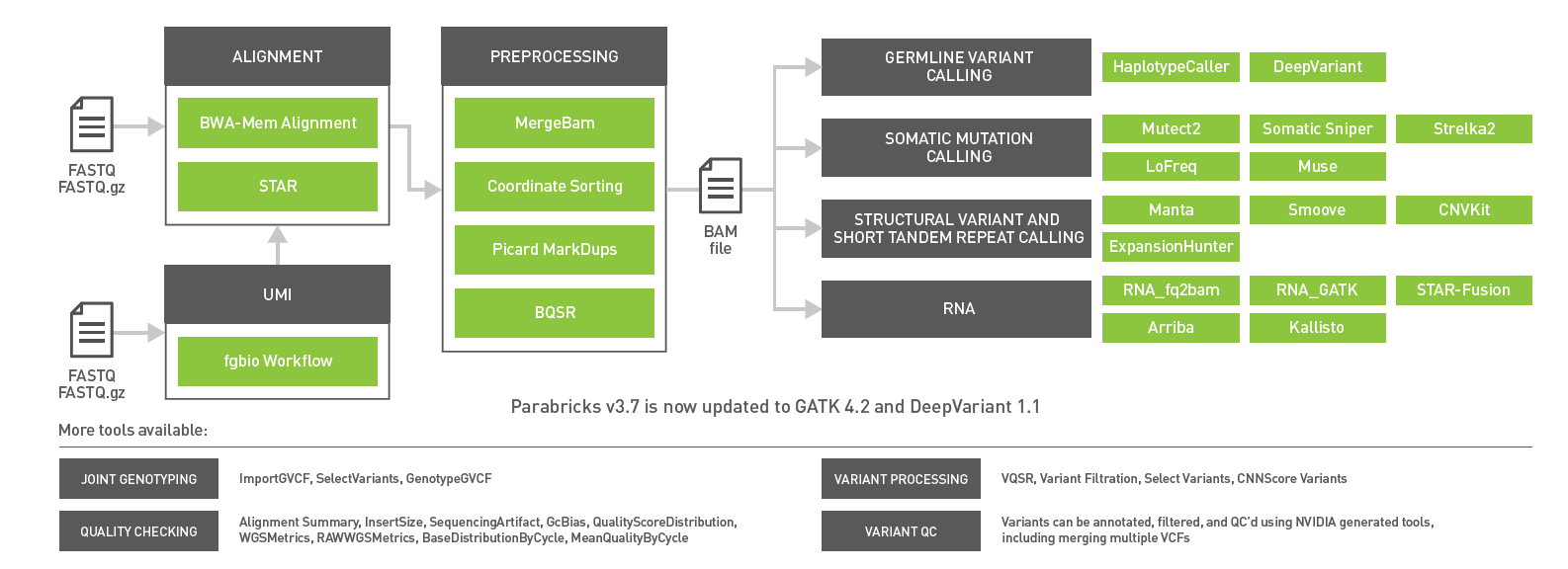
由 GPU 驱动的 NVIDIA Clara Parabricks v3 的最新版本。 7 包括对种系变体调用者的更新、对 RNA 序列管道的增强支持以及基因面板的优化工作流程。现在有超过 50 种工具, Clara Parabricks 为临床和研究工作流程中的基因面板、外显子组和基因组提供准确和加速的基因组分析。
迄今为止, Clara Parabricks 已经证明,与基于 CPU 的环境相比,用于全基因组工作流程( 22 分钟内完成端到端分析)和外显子组工作流程( 4 分钟内完成端到端分析)的最先进生物信息学工具的速度提高了 60 倍。大规模测序项目和其他全基因组研究能够在单个 DGX 服务器上每天分析 60 多个基因组,同时降低了相关成本,产生了比以往任何时候都有用的见解。
包括 泰国国家生物银行 、 人类基因组中心与东京大学 、 基因组学研究院 、 圣路易斯华盛顿大学 、 Regeneron 遗传学中心和英国生物银行 和 斯坦福大学 Euan Ashley 的实验室 在内的许多组织正在使用 Clara Parabricks 进行快速基因组和外显子组分析,以用于大人口项目、危重患者以及其他癌症和遗传病项目。他们的工作旨在准确、快速地识别致病变异,与加速的下一代测序和加速的基因组分析保持同步。
NVIDIA Clara Parabricks v3.7 概述
- 通过支持独特的分子标识符( UMIs ),也称为分子条形码,加快和简化基因面板工作流程。
- 使用第二个基因融合调用者 Arriba 和 RNA-Seq 定量工具 Kallisto 对转录组工作流的 RNA-Seq 支持。
- 用 ExpansionHunter 检测短串联重复序列( STR )。
- 集成最新版本的生殖系调用者 DeepVariant v1 。 1 和 GATK v4 。 2 为单倍型。
- 一个 10 倍加速的 BAM2FASTQ 工具,用于将存储为 BAM 或 CRAM 文件的存档数据转换回 FASTQ 。数据集可以根据新的和改进的引用进行更新。

Clara Parabricks 3.7 加速和简化了基因面板分析
尽管全基因组测序( WGS )由于大规模的人群计划而不断增长,但基因面板仍然主导着临床基因组分析。随着时间成为临床护理中最重要的因素之一,加速和简化基因小组工作流程对于临床测序中心至关重要。通过进一步减少与基因面板相关的分析瓶颈,这些测序中心可以更快地向临床医生返回结果,提高患者的生活质量。
用于基因检测的癌症样本通常来自实体肿瘤或患者血液(液体活组织检查)中的无细胞 DNA 。与 WGS 的发现工作相比,基因小组狭隘地专注于识别已知基因中的基因变体,这些基因要么会导致疾病,要么可以通过特定疗法进行靶向治疗。
遗传性疾病的基因面板通常测序覆盖率为 100 倍,而癌症测序的基因面板测序深度要高得多,液体活检样本的测序深度高达 1000 倍。检测与癌症相关的低频率体细胞突变需要更高的覆盖率。
为了提高这些基因面板的检测极限,使用了分子条形码或 UMI ,因为它们显著降低了背景噪声。这一检测极限适用于液体活组织检查,可以包括数以万计的检测范围,并与 UMIs 相结合,以识别血液中循环的大海捞针体细胞突变。高深度基因面板测序可以在更多测序读取所需的处理中重新引入计算瓶颈。
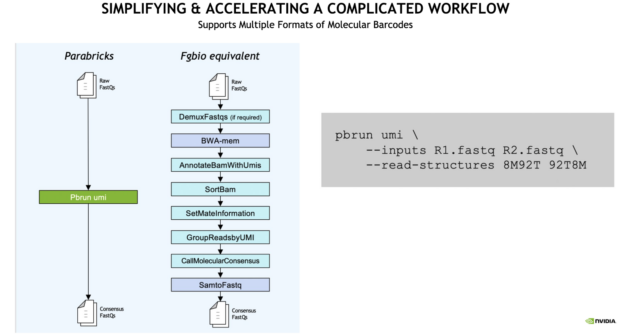
使用 Clara Parabricks , UMI 基因面板现在可以比传统工作流程快 10 倍,在不到一小时的时间内产生结果。
分析工作流程也得到了简化。从 raw FASTQ 到 consensus FASTQ ,与传统的 Fulcrum Genomics ( Fgbio )等价物相比,单个命令行运行多个输入,如下面的示例所示。

Arriba 和 Kallisto 的 RNA Seq 支持
正如基因面板对于癌症测序分析很重要一样, RNA-Seq 工作流程对于转录组分析也很重要。除了恒星融合, Clara Parabricks v3 。 7 现在包括 Arriba ,一种基于恒星的融合检测算法 RNA 序列比对器。基因融合是两个不同的基因由于染色体的巨大改变而结合在一起,与从白血病到实体瘤等多种不同类型的癌症有关。
Arriba 还可以检测病毒整合位点、内部串联重复、整外显子重复、环状 RNA 、涉及免疫球蛋白/ T 细胞受体位点的增强子劫持事件以及内含子或基因间区域的断点。
Clara Parabricks v3 。 7 还结合了 Kallisto ,这是一种基于伪比对的快速 RNA 序列量化工具,可从批量或单细胞 RNA 序列数据集中识别转录物丰度(基于测序读取计数的 aka 基因表达水平)。 RNA 序列数据的比对是 RNA 序列分析工作流程的第一步。 Clara Parabricks 3.7 提供了转录本定量、读取比对和融合调用的工具,现在提供了一整套支持多个 RNA 序列工作流的工具。
用 ExpansionHunter 进行短串联重复检测
为了支持从短读测序数据中对短串联重复序列( STR )进行基因分型,在 Parabricks v3 中添加了 ExpansionHunter 支持。 STR ,也称为微卫星,在人类基因组中普遍存在。这些非编码 DNA 区域具有类似手风琴的 DNA 延伸,包含长度在 2 到 7 个核苷酸之间的核心重复单元,串联重复,最多几十次。
STR 在遗传图谱构建、基因定位、遗传连锁分析、个体鉴定、亲子鉴定、群体遗传学和疾病诊断等应用中非常有用。
人类基因组中有许多区域由这些重复序列组成,它们的重复次数会增加,从而导致疾病。脆性 X 综合征、肌萎缩侧索硬化症和亨廷顿氏病是常见的重复相关疾病。
ExpansionHunter 旨在通过对 BAM / CRAM 文件执行有针对性的搜索来估计重复序列的大小,以便对每个 STR 中完全包含的跨、侧翼和侧翼读取进行排序。
添加体细胞呼叫者并支持归档数据
2021 以前的版本 Clara Parabricks 带来了一系列新的工具,最重要的是增加了五个躯体学呼叫者 MuSE 、 LoFReq 、 StRekA2 、 MutuSc2 和体细胞狙击手,用于全面的癌症基因组分析。

此外,还添加了一些工具,以便在删除原始 FASTQ 文件以节省存储空间的情况下利用归档数据。 BAM2FASTQ 是 GATK Sam2fastq 的加速版本,它将现有 BAM 或 CRAM 文件转换为 FASTQ 文件。这允许用户将测序读数重新调整到一个新的参考基因组,这将允许调用更多的变体,同时为研究人员提供将所有数据标准化为同一参考的能力。使用现有的 fq2bam 工具, Clara Parabricks 可以在不到 2 小时的时间内将读取从一个参照物重新排列到另一个参照物,从而获得 30 倍的全基因组。

- 使用 免费提供 90 天试用评估许可证 为您的种系、癌症和 RNA 序列分析工作流试用 GPU 加速的 NVIDIA Clara Parabricks 基因组分析工具。
- 通道 NVIDIA Clara 抛物面 通过 AWS Marketplace 在本地或云中进行。
- 阅读更多关于 Parabricks v3 的信息。 7 释放 .